SAILアミノ酸/メチル選択標識アミノ酸・芳香族標識アミノ酸


SAIL法により拡がるタンパク質NMRの未来
甲斐荘正恒
東京都立大学
名誉教授
「Biomolecular NMR」カタログ(大陽日酸 2015) 寄稿文
SAIL法により拡がるタンパク質NMRの未来

甲斐荘正恒
東京都立大学
名誉教授
はじめに
X線結晶構造解析に代表されるタンパク質の立体構造決定手法は急速な発展を遂げ、以前は想像もできなかったような巨大、且つ複雑なタンパク質複合体の精密な立体構造が次々と解明されている。しかしながら、結晶中で整然と静止した状態における立体構造情報のみでは、実際に生物機能を果たすタンパク質の真の姿を理解するには十分ではない。タンパク質の立体構造と生物機能の関連をより深く理解するためには、
生体内において各タンパク質が存在する環境にできる限り近い状態で、それらの立体構造・動態・相互作用を総合的に解明する手段が不可欠となる。数々の困難にもかかわらず、タンパク質のNMR研究が人々をひきつける理由がここにある。半世紀に渡るたゆみない努力の結果、TROSY(Transverse Relaxation Optimized SpectroscopY)法をはじめとして様々な技術が続々と開発され、NMR法の最大の弱点とされてきた分子量限界の大幅な拡張が実現しつつある。ここで紹介するSAIL(Stereo-Array Isotope Labeling;立体整列同位体標識)法もそのような成果の一つであるが、他の手法にはみられない際立った特徴がある。即ち、従来の技術開発では、高分子量タンパク質の主鎖アミド(15N1H)やIle、 Leu、Val等のメチル基(13C1H3)等の
一握りのNMR情報を入手するために、他の原子団に由来するNMR情報の取得を犠牲にすることを前提としてきた。我々の開発したSAIL法は、安定同位体標識パターンを徹底的に最適化したアミノ酸(SAILアミノ酸)を合成し、それらで標識したタンパク質(SAILタンパク質)を用いることにより、あらゆる原子団由来のNMRシグナルを余すことなく観測しようとする技術である。SAIL標識した高分子量タンパク質のNMRスペクトルは著しく簡略化される一方、測定感度も大きく改善される。究極的なTROSY 法とも云えるSAIL法は、これまで入手することが困難とされてきた原子団に関する様々な立体構造や動態に関する新たな情報を通じて、高分子量タンパク質複合体の生物機能に迫るNMR技術として今後の発展が期待される1 )。
タンパク質NMR研究における安定同位体利用技術
タンパク質の主要な構成元素、水素、炭素、窒素、酸素のうちで高分解能NMRシグナルが観測できる安定同位元素は1H13C、15Nの3種類である。水素(1H)を除きこれらの元素は天然に存在する割合(天然存在比)が低いためにNMR観測には高濃度の同位体標識が必要となる。低分子化合物ならば、1H-NMRスペクトルから必要十分な構造情報が得られるが、20種類のアミノ酸残基のみから構成されるタンパク質のNMRスペクトルで状況は大きく異なる。即ち、タンパク質の1H-NMRスペクトルでは、近接した化学シフトを持つ膨大な1H-シグナルが重なり合い、更に1H-1H間の双極子相互作用による線幅の拡がりも加わるため、それらの解析には多大な困難が伴う。多次元NMR法に代表されるタンパク質構造解析技術の発展の基盤は、
このような困難を軽減するために追求された、安定同位体標識技術の高度化に向けた膨大な努力から形成されている2 )。安定同位体標識法は、立体構造や生物機能への影響を最小限に抑えつつ、タンパク質NMRスペクトル上の特性を大きく変えることができる手法である。例えば、天然に存在する炭素と窒素の殆どは同位元素12C、14N であるが、前者は核スピンを持たないためNMR測定にかからず、後者は核スピンが1のために高分解能NMR測定には不向きである。しかしながら、これらの元素を核スピン1/2を持つ安定同位体13C、15Nに置換することにより、タンパク質の骨格元素である炭素、窒素を溶液NMR法に組み込むことが可能となる。その結果、1H, 13C,15Nを含めた多核種多次元NMR測定法が成立し、高分子量タンパク質のNMRスペクトル解析は著しく容易となる。更に、水素の同位元素である重水素(2H)でタンパク質を標識すれば、
1H-NMRスペクトルは大幅に簡略化されると同時に、1H-密度の低下に伴い重水素置換されずに残った1H-シグナルのNMRシグナルの線幅は大幅に狭まり、延いては測定感度の著しい増加という恩恵がもたらされる。この結果、13C /15N-二重標識試料を用いた手法では、構造決定が可能な分子量上限が25 kDa程度であったものが、2H/13C/15N-三重標識試料の利用による更なる分子量上限の拡張が可能となる。しかしながら、三重標識試料を用いて、タンパク質中の必要な部位の1H-NMR情報を得るためには、その部分の重水素化だけは選択的に避ける必要がある。試料調製の簡便さから、適当な重水素濃度に調整した重水中(例えば、60-80%程度)で、“ランダム” 重水素化を施した三重標識試料を用い“残余1H”を観測する手法が試みられた。
しかしながら、このような手法では重水素化の位置や濃度を制御できないために、無数の同位体異性体(isotopomer)が混在することは避けられない。このような根本的な欠陥のために、現在ではほとんど利用されていない。
SAIL- 立体整列同位体標識法の誕生
我々は、このような状況を根本的に解決するには、重水素化を含めた安定同位体標識技術をその限界まで高め、立体選択性を含めて標識位置を完全に制御したタンパク質試料を調製できるならば、得られる構造情報の精度を犠牲にせず、長い間の懸案事項であった分子量限界を大幅に拡張できると考えた。これが、SAIL法の基本的な発想である1 )。このために、下記に列挙した原則に従い、タンパク質NMR解析に適した同位体標識パターンを持つアミノ酸を合成した。図1に示した標識パターンは、SAIL法の開発当初のもの(“第一世代” SAILアミノ酸)であるが、その後現在に至るまで多種多様な改良型SAILアミノ酸が開発され、様々な構造・動態情報の取得に利用されている。
(1) アミノ酸に含まれる全てのメチレン基上のプロキラル水素のうち、一方のみを立体選択的に重水素化する。
(2) アミノ酸に含まれるメチル基を13CHD2標識する。また、Leu、Val のプロキラルメチル基のうち、一方を立体選択的に12CD3、他方を13CHD2とする(現在は経済性を考えて13CHD2の代わりに13CH3とするパターンを主に利用)。
(4) 芳香族アミノ酸、Phe、Tyr、Trp の芳香環においては、観測対象とする炭素と水素のみ1H-13C とし、他は 12CDとする。
(5) 水素を持つ炭素は全て13C、窒素は全て15Nとすることにより、13C-13C、13C-15Nスピン結合を経由し側鎖原子団の帰属を可能とする。
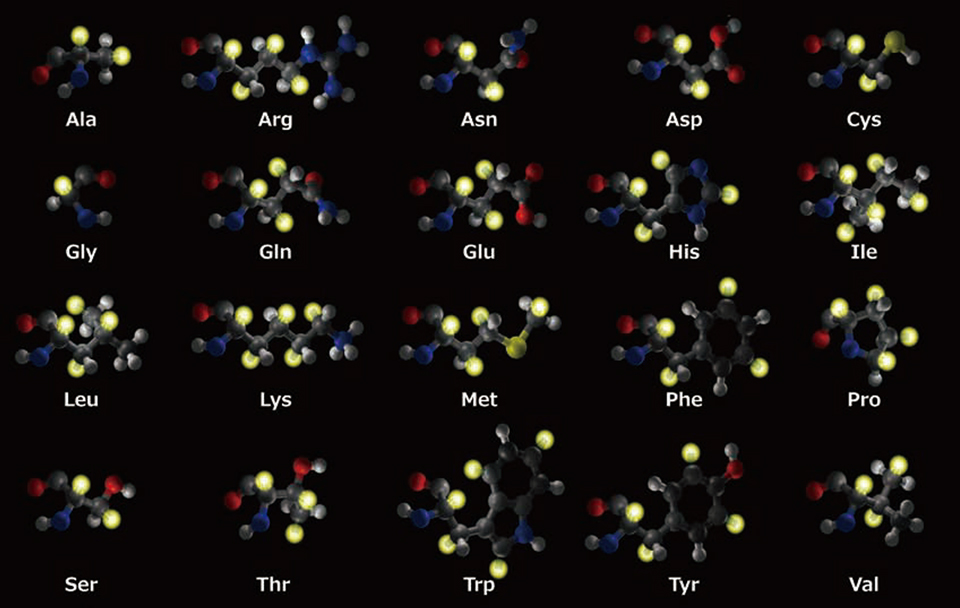
図1 タンパク質を構成する20 種類の“ 第一世代”SAIL アミノ酸の化学構造. 1Hとして残した側鎖水素はベージュ, 重水素化したものは灰色で表示. その他、窒素は青, 炭素は黒, 酸素は赤, 硫黄は黄色で表示してある. また, 1Hの結合した炭素は全て13C, 窒素は全て15Nで標識してある(. グラフィックスはKimmo Pääkkönen博士作成)
以上の原則に従った同位体標識パターンを持つタンパク質を構成する20種類のアミノ酸を“第一世代”のSAILアミノ酸と呼んでいるが、それらの合成が完了するには10年余の歳月を要した。SAILアミノ酸はプロキラルなメチレン、メチル基は立体選択的に同位体標識されているために、NMRによる構造精度に大きく影響する立体帰属に関する不確実性は全く存在しないため、重水素化により失われる情報量の減少を補って余りある情報の質的向上が得られる。もう一つのSAIL法の特記すべき特徴は、SAILアミノ酸のみから構成されるタンパク質(SAILタンパク質)は、13C,15N-二重標識タンパク質と比較して、タンパク質全体の水素密度は50-60%近く減少するにも関わらず、重水素化部位以外の水素(残余水素)は完全に保存される点である。従って、重水素化の位置・立体化学を制御できないランダム重水素化法では問題となる、
同じ標識パターンを持つ分子が理論的には存在し得ないほど無数に存在する同位体異性体は、SAILタンパク質では只一種類の同位体異性体に集約される。このような全く新しい概念の安定同位体標識技術を立体整列同位体標識(Stereo-array isotope labeling;SAIL)法と名付けた理由がここにある。SAILアミノ酸には合成が困難なものが幾つも含まれており、従ってこのような貴重なアミノ酸の効率的利用がSAIL法の実用化にとっての鍵となる。このためには、SAILアミノ酸の安定同位体標識パターンを保ったまま目的とするタンパク質に効率良く取り込ませることのできる大腸菌無細胞合成システムが最も有効である。このようにしてSAILアミノ酸のみからなるSAILタンパク質の調製が実現した。SAILタンパク質においては、全てのプロキラル基の一方が立体特異的に重水素化されているため、観測されるプロキラル基の立体帰属は全て確定している。
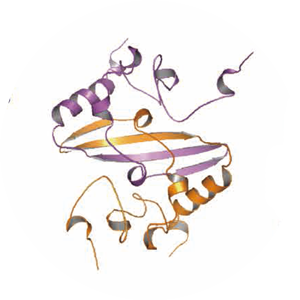
このために、従来の立体帰属が不確実な場合に利用せざるを得ない擬原子(pseudoatom)近似が不要となるために、遥かに高精度(precise)、且つ正確(accurate)な立体構造が得られる1 , 2 )。以下に、その一例としてSARS コロナウィルス C末側RNA結合ドメインの溶液構造決定を紹介する3 )。
SAIL法によるSARS コロナウィルス C末側ドメインの立体構造決定
数年前、世界を震撼とさせた重症呼吸器性症候群(SARS)の発症原因となるコロナウィルス(SARS-CoV)は、ヌクレオキャプシドと呼ばれるタンパク質とRNAからなる集合体を形成している。S A R S - C o V のキャプシドタンパク質は、422 残基のアミノ酸からなり、そのN末端側およびC末端側の2ヶ所に構造ドメインを持つ。
両ドメインともRNA 結合能を持つが、C末端側ドメイン(C-terminal domain; CTD)は、タンパク質の二量化形成の役割も担う。そのため、本研究の共同研究先の台湾中央研究院においてはSARS-CoV CTD の立体構造解明が重要な緊急課題となっていた。しかしながら、シグナルの縮重、及び線幅の拡がりに阻まれ、従来のNMR手法ではトポロジー構造の決定に留まっていた。SAIL法を用いた共同研究が持ちかけられたのはこのような事情による。SARS-CoV CTD の構造決定は数週間で完了したが、SAIL法の適用によりもたらされた最も大きなNMRスペクトル上の改善点は、芳香族領域のNMRスペクトルの劇的な向上にあった。図2-aに示すように、通常の[U-13C,15N]-標識体では、芳香環領域の1H-13C HSQCスペクトルは複雑なスピン系のために、有用な構造情報が全く得られなかった。
一方、SAIL-標識体の同じ芳香環領域では、極めて良質の1H-13C HSQCスペクトルが容易に得られた(図2-b)。この芳香族領域のNMRスペクトルの質的改善がSARS-CoV CTD の迅速な精密構造決定の決め手となった。S A R S - C o V C T D は、4本鎖から構成される逆平行βシート上に、α へリックスが位置するいわゆる“domain-swapped dimer” 構造を形成しており(図2-c)、このNMR構造は後に決定された結晶構造とも良く一致した。SAILタンパク質を用いた核酸滴定実験等の相互作用解析も実施し、SARSの薬剤開発に重要な基礎的知見が得られた3 )。幸いなことに、SARSは終息後、現在に至るまで大きな流行が報告されていない。
SAIL法の展望
本稿ではSAIL法の原理とNMR構造決定への応用に関して簡単に解説したが、現在も高分子量タンパク質への応用、タンパク質動態の研究手法の開発等に向けSAIL法の応用分野の開発が活発に進められている。SAILアミノ酸合成に関連した不斉有機合成技術の高度化、及び標識アミノ酸を用いて効率良く目的タンパク質を標識する無細胞系・細胞系発現技術は過去数年間で大きく発展し、新たなNMR研究技術の開発基盤を提供している。革新的安定同位体利用NMR技術であるSAIL法の更なる開発と普及は、生体系NMR研究の活性化に大きく寄与するだけではなく、我が国の生命科学基盤技術への国際貢献という点からも重要な課題であろう。
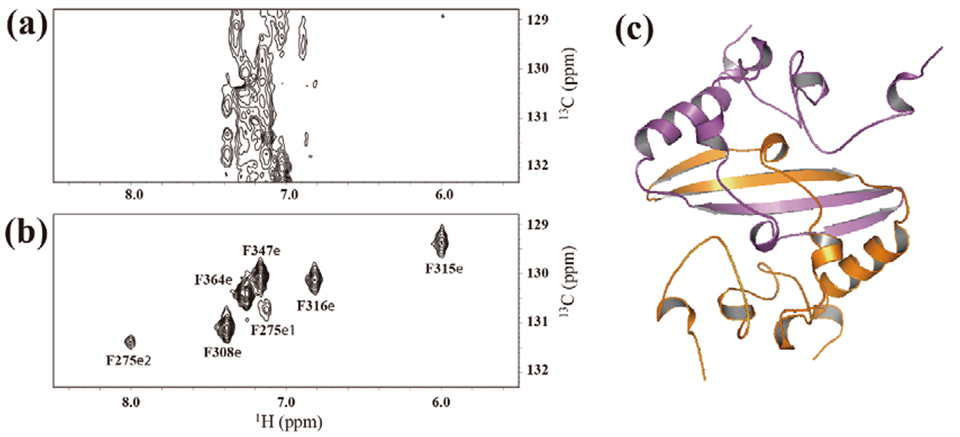
図2 芳香環領域の 1H-13C HSQC スペクトル(: a)[U-13C,15N]-SARS CoV CTD(, b)SAIL SARS-CoV CTD(. c)SARS-CoV のNMR構造のリボン図. SAIL法ではCTDドメインに含まれる7 残基Pheの6 残基の13Cε-1Hεのシグナルが明瞭に観測された. なお, F275 は芳香環の反転速度が遅いためにε1, ε2 が分裂して観測され, F 287 のε- シグナルは反転によるシグナルの広幅化により観測されない. このような芳香環の反転速度に関する情報はSAIL法がタンパク質動態の研究に有用であることを示している.
参考文献
1)“Optimal Isotope Labelling for NMR Protein Structure Determinations”, M. Kainosho, T. Torizawa, Y. Iwashita, T. Terauchi, A. M. Ono, and P. Guentert, Nature,440, 52-57 (2006); “SAIL- Stereo-array isotope labeling”, M. and P. Güntert, Quarterly Reviews of Biophysics, 42, 247-300 (2009).
2)“Recent Developments in Stable-Isotope-Aided Methods for Protein NMR Spectroscopy”, S.-y. Ohki and M. Kainosho, Modern Magnetic Resonance, 211-218 (2006); “Stable Isotope Labeling Methods for Protein NMR Spectroscopy”, S.-y. Ohki and M. Kainosho, Progress in Nuclear Magnetic Resonance Spectroscopy, 53, 208-226 (2008).
3)“Solution Structure of the C-terminal Dimerization Domain of SARS Coronavirus Nucleocapsid Protein Solved by the SAILNMR
Method”, M. Takeda, C.-k.Chang, T.Ikeya, P. Güntert, Y.-h. Chang, Y.-l. Hsu, T.-h. Huang, and M. Kainosho, J. Mol Biol., 380, 608-622 (2008).


イノベーションユニット
SI事業部
045-872-1823
※月~金曜日
9:00~17:40
045-872-1825
Isotope.TNS@tn-sanso.co.jp
〒220-8561
神奈川県
横浜市西区
みなとみらい4-6-2
みなとみらい
グランドセントラルタワー 7F
大陽日酸㈱
みなとみらい事業所内

高圧ガス販売事業(第1種、第2種)
毒物劇物一般販売業 / 毒物劇物輸入業
医療をはじめ、研究から産業まで広くご利用いただける安定同位体試薬をご提供いたします。
Copyright © 2021. TAIYO NIPPON SANSO Corporation. All Rights Reserved.